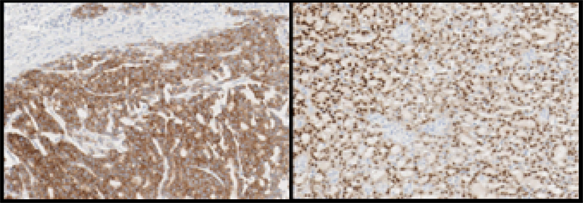
Sólo pruebas sensibles y específicas pueden detectar de manera fiable las fusiones génicas NTRK y ROS1 1-3
- Existen múltiples pruebas moleculares disponibles para detectar fusiones de genes, pero algunas son más específicas que otras1,2,4-11
- La secuenciación de última generación (NGS) tiene alta sensibilidad, exactitud y rendimiento necesarios para evaluar todas las fusiones de genes9-16
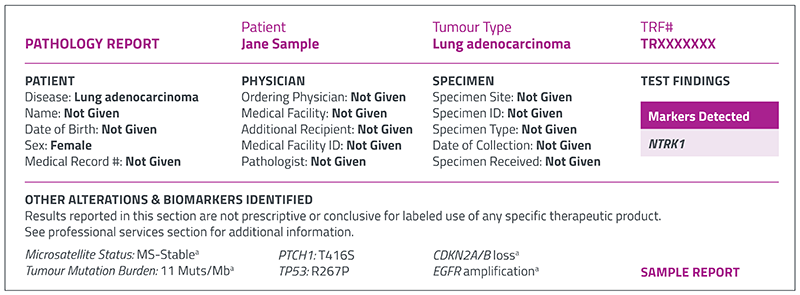
Tecnologías disponibles para la detección de fusiones génicas
Una prueba molecular apropiada puede ayudar a descubrir fusiones génicas.4,5,18 Tu patólogo puede ayudar a decidir cuál es la mejor opción de prueba para cada paciente.
Notas a pie de página:
FISH, hibridización de fluorescencia in situ; IHC, inmunohistoquímica; NGS, secuenciación de nueva generación; CPCNP, cáncer de pulmón de células no pequeñas; RT-PCR, reacción de cadena de polimerasa de transcripción reversa.
- 1
Su D, et al. J Exp Clin Cancer Res 2017;36:1–12.
- 2
Hechtman JF, et al. Am J Surg Pathol 2016;41:1547–1551.
- 3
Kumar-Sinha C, Kalyana-Sundaram S, Chinnaiyan AM. Genome Med 2015;7:1–18.
- 4
Vaishnavi A, et al. Cancer Discov 2015;5:25–34.
- 5
Murphy DA, et al. Appl Immunohistochem Mol Morphol 2017;25:513–523.
- 6
Stack EC, et al. Methods 2014;70:46–58.
- 7
Knezevich SR, et al. Nat Genet 1998;18:184–187.
- 8
Naidoo J, Drilon A. Am J Hematol Onco 2014;10:4–11.
- 9
International Association for the Study of Lung Cancer. IASLC Atlas of ALK and ROS1 Testing in Lung Cancer. Available at: https://www.iaslc.org/sites/default/files/wysiwyg-assets/alk-ros1_atlas_lowres.pdf (Accessed April 2019).
- 10
Shan L, et al. PLoS One 2015;10:e0120422.
- 11
Bubendorf L, et al. Virchows Arch 2016;469:489–503.
- 12
Cao B, et al. Onco Targets Ther 2016;31:131–138.
- 13
National Comprehensive Cancer Network. NCCN Clinical Practice Guidelines in Oncology (NCCN Guidelines). Non-Small Cell Lung Cancer. V.5.2018, 2018. Available at: www.nccn.org/professionals/physician_gls/recently_updated.aspx (Accessed April 2019).
- 14
Zheng Z, et al. Nat Med 2014;20:1479–1484.
- 15
Drilon A, et al. Clin Cancer Res 2015;21:3631–3639.
- 16
Grada A, Weinbrecht K. J Invest Dermatol 2013;133:e11.
- 17
Horak P, et al. ESMO Open 2016;1:e000094.
- 18
Penault-Llorca F, et al. J Clin Pathol 2019;72:460–467.
- 19
Kummar S, Lassen UN. Target Oncol 2018;13:545–556.
- 20
VENTANA pan-TRK (EPR17341) Assay: Package Insert 1017533EN Rev A.
- 21
Rossi G, et al. Lung Cancer (Auckl) 2017;8:45–55.
- 22
Ali G, et al. Arch Pathol Lab Med 2018;142:480–489.